Hầu hết người mới bắt đầu trong Học máy đều bắt đầu với việc học các kỹ thuật Học có giám sát như phân loại và hồi quy. Tuy nhiên, một trong những mô hình quan trọng nhất trong Machine Learning là Reinforcement Learning (RL) có thể giải quyết nhiều nhiệm vụ khó khăn. Đây là một khía cạnh của Học máy trong đó tác nhân học cách cư xử trong một môi trường, bằng cách thực hiện các hành động nhất định và quan sát phần thưởng (kết quả) mà nó nhận được từ những hành động đó.
Trong Học tập củng cố, chúng ta cung cấp cho máy một vài đầu vào và hành động, sau đó thưởng cho chúng dựa trên kết quả đầu ra. Tối đa hóa phần thưởng là mục tiêu cuối cùng. Nó giống như một đứa trẻ nhỏ không biết gì lúc đầu bị bỏ lại một mình trong một môi trường và sau đó sau khi hiểu biết một chút có xu hướng tự mình biết mọi thứ xung quanh.
Ở đây, chúng ta sẽ xây dựng một thuật toán dựa trên sự đột biến gen của một quần thể khi bị virus tấn công. Trong thế hệ đầu tiên của dân số chúng ta chỉ có một số người khỏe mạnh nhất có thể sống sót, trong khi các thế hệ trôi qua, các thế hệ mới sẽ chống lại virus mạnh hơn nhiều so với tổ tiên của chúng. Nó là một thuật toán cơ bản chỉ đưa ra ý tưởng về cách những thứ này hoạt động. Bất kỳ ai có kiến thức cơ bản về python và một số thư viện như numpy, matplotlib, v.v. đều có thể dễ dàng hiểu mã này. Đây chỉ là phần giới thiệu và cung cấp kiến thức ở mức bề mặt về Học tăng cường.
1. Thuật toán di truyền
Thư viện được sử dụng:
- numpy: chúng ta sẽ sử dụng mảng numpy và các chức năng tính toán cơ bản khác từ thư viện này
- matplotlib: chúng ta sẽ sử dụng chức năng matplotlib.pyplot để vẽ biểu đồ giúp bạn hiểu rõ về thuật toán.
Trong chương trình này, chúng ta sẽ xác định 3 chức năng chính để tạo ra thế hệ tiếp theo của quần thể mạnh hơn về mặt di truyền so với thế hệ trước.
Ba chức năng chính được sử dụng là:
- Nơi chứa: Điều này được sử dụng để tạo tổng thể và sau đó thêm nó vào danh sách. Hàm này chấp nhận các thuộc tính như số lượng đối tượng trong tổng thể và kích thước của nó.
- Giảm: Chức năng này được sử dụng để giảm kích thước của quần thể và chỉ cho phép 100 con khỏe mạnh nhất sống sót. Những con phù hợp này sẽ là những con được sử dụng để tạo ra thế hệ tiếp theo của quần thể.
- chéo: Chức năng này được sử dụng cho quá trình lai tạo giữa những con còn lại để tạo ra một thế hệ quần thể hoàn toàn mới sẽ miễn dịch hơn nhiều đối với vi rút.
Dưới đây là cách thực hiện –
# -----------------------------------------------------------
#Cafedev.vn - Kênh thông tin IT hàng đầu Việt Nam
#@author cafedevn
#Contact: cafedevn@gmail.com
#Fanpage: https://www.facebook.com/cafedevn
#Group: https://www.facebook.com/groups/cafedev.vn/
#Instagram: https://instagram.com/cafedevn
#Twitter: https://twitter.com/CafedeVn
#Linkedin: https://www.linkedin.com/in/cafe-dev-407054199/
#Pinterest: https://www.pinterest.com/cafedevvn/
#YouTube: https://www.youtube.com/channel/UCE7zpY_SlHGEgo67pHxqIoA/
# -----------------------------------------------------------
import numpy as np
import matplotlib.pyplot as plt
# specifying the size for each and
# every matplotlib plot globally
plt.rcParams['figure.figsize'] = [8, 6]
# defining list objects with range of the graph
x1_range = [-100, 100]
x2_range = [-100, 100]
# empty list object to store the population
population = []
# this function is used to generate the population
# and appending it to the population list defined above
# it takes the attributes as no. of features in a
# population and size that we have in it
def populate(features, size = 1000):
# here we are defining the coordinate
# for each entity in a population
initial = []
for _ in range(size):
entity = []
for feature in features:
# this * feature variable unpacks a list
# or tuple into position arguments.
val = np.random.randint(*feature)
entity.append(val)
initial.append(entity)
return np.array(initial)
# defining the virus in the form of numpy array
virus = np.array([5, 5])
# only the 100 fit ones will survive in this one
def fitness(population, virus, size = 100):
scores = []
# enumerate also provides the index as for the iterator
for index, entity in enumerate(population):
score = np.sum((entity-virus)**2)
scores.append((score, index))
scores = sorted(scores)[:size]
return np.array(scores)[:, 1]
# this function is used to plot the graph
def draw(population, virus):
plt.xlim((-100, 100))
plt.ylim((-100, 100))
plt.scatter(population[:, 0], population[:, 1], c ='green', s = 12)
plt.scatter(virus[0], virus[1], c ='red', s = 60)
def reduction(population, virus, size = 100):
# only the index of the fittest ones
# is returned in sorted format
fittest = fitness(population, virus, size)
new_pop = []
for item in fittest:
new_pop.append(population[item])
return np.array(new_pop)
# cross mutation in order to generate the next generation
# of the population which will be more immune to virus than previous
def cross(population, size = 1000):
new_pop = []
for _ in range(size):
p = population[np.random.randint(0, len(population))]
m = population[np.random.randint(0, len(population))]
# we are only considering half of each
# without considering random selection
entity = []
entity.append(*p[:len(p)//2])
entity.append(*m[len(m)//2:])
new_pop.append(entity)
return np.array(new_pop)
# generating and adding the random features to
# the entity so that it looks more distributed
def mutate(population):
return population + np.random.randint(-10, 10, 2000).reshape(1000, 2)
# the complete cycle of the above steps
population = populate([x1_range, x2_range], 1000)
# gens is the number of generation
def cycle(population, virus, gens = 1):
# if we change the value of gens, we'll get
# next and genetically more powerful generation
# of the population
for _ in range(gens):
population = reduction(population, virus, 100)
population = cross(population, 1000)
population = mutate(population)
return population
population = cycle(population, virus)
draw(population, virus) Kết quả
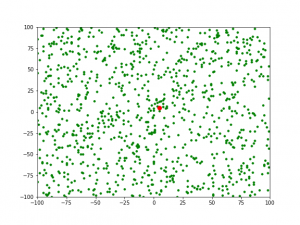
1) Đối với thế hệ 1, khi gens = 0
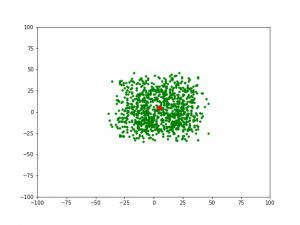
2) Đối với thế hệ 2, khi gens = 1
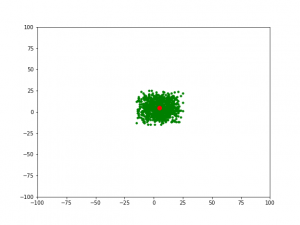
3) Đối với thế hệ 3, khi gens = 2
Cài ứng dụng cafedev để dễ dàng cập nhật tin và học lập trình mọi lúc mọi nơi tại đây.
Nguồn và Tài liệu tiếng anh tham khảo:
Tài liệu từ cafedev:
- Full series tự học Python từ cơ bản tới nâng cao tại đây nha.
- Tự học ML bằng Python từ cơ bản tới nâng cao.
- Ebook về python tại đây.
- Các series tự học lập trình MIỄN PHÍ khác
- Nơi liên hệ hợp tác hoặc quảng cáo cùng Cafedevn tại đây.
Nếu bạn thấy hay và hữu ích, bạn có thể tham gia các kênh sau của cafedev để nhận được nhiều hơn nữa:
Chào thân ái và quyết thắng!








![[Tự học C++] Số dấu phẩy động(float, double,…) trong C++](https://cafedev.vn/wp-content/uploads/2019/12/cafedevn_c_develoment-100x70.jpg)